Nous avons vu comment chercher une sous-chaîne en utilisant la programmation dynamique. Nous étudierons plus généralement (c’est à dire en dehors du cadre de la programmation dynamique) la recherche d’une chaîne dans un texte et en particulier l’algorithme de Boyer et Moore.
Cette recherche a de nombreuses applications et en particulier dans la génétique.
Commençons par étudier une manière naïve, c'est-à-dire avec un raisonnement simple, de chercher une sous-chaîne dans une chaîne.
1) Recherche naïve¶
Les molécules d’ADN sont composées de deux brins antiparallèles enroulés l’un autour de l’autre.

On peut alors représenter l’information génétique par de très longues chaînes. On parlera de séquences ADN, où les lettres correspondent à chacunes des bases des acides nucléiques. Il existe une convention qui correspond au sens de parcours des brins d’ADN, ce qui dépasse largement le cadre de ce cours.
A correspond à l’adénine, C à la cytosine, G à la guanine, et T à la thymine. Supposons que l’on cherche un certain enchaînement dans une certaine séquence.
Il existe des bases de données libres qui permettent d’accéder à des séquences ADN. Pour rendre notre exemple plus concret, nous allons utiliser le génome d’un variant du Coronavirus que l’on peut récupérer sur le site du NIH (l’équivalent des CHU mais aux états-unis) : https://www.ncbi.nlm.nih.gov/nuccore/NC_045512.2?report=fasta
Le format de fichier FASTA est un format très utilisé dans le domaine du séquençage ADN. On remarquera que l’ARN a été traduit en ADN (d’où la présence de Thymine et l’absence d’Uracile).
Supposons que l’on cherche la séquence suivante : TTCACCGA qui correspond au début d’une structure en tige-boucle aussi appelée structure en épingle à cheveux à cause de sa forme partiulière :
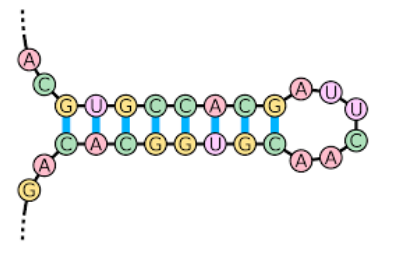
Pour que notre exemple soit plus facile à expliquer, nous allons prendre uniquement une petite partie du génome du virus.
Supposons que je cherche ce “motif” dans cette section là du génome :
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATCAGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- On place notre motif au niveau des huit premières lettres de la chaîne qui correspond à l’extrait du génome.
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
-
On compare chacune des lettres en commençant avec la première lettre à gauche. La première lettre du motif que l’on recherche ne correspond pas avec la première lettre de la chaîne.
-
On décale alors le motif d’une lettre vers la droite.
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- La première lettre du motif correspond cette fois-ci à la première lettre T de la chaîne. On compare alors la deuxième lettre du motif à la deuxième lettre de la chaîne. T ne correspond pas à G, on décale alors à nouveau le motif d’un cran à droite.
On voit que cet algorithme peut demander beaucoup de comparaisons et de décalages. Nous allons étudier un algorithme qui sera plus efficace pour résoudre ce problème : l’algorithme de Boyer-Moore. Nous étudierons une version simplifiée de cet algorithme.
2) Algorithme de Boyer-Moore simplifié (dit de Horspool)¶
2.1) Premier exemple¶
L’algorithme de Boyer-Moore va effectuer un traitement sur le motif, ce qui veut dire qu’il va s’appuyer sur l'information contenue dans le motif (les lettres qu’il contient) qui composent le motif pour ne pas avoir à comparer les lettres une à une comme on l’a fait dans notre algorithme naïf.
Ce traitement consite à calculer les décalages nécessaires lorsqu'une lettre qui se trouve dans le motif ne correspond pas avec la partie du texte sur laquelle on effectue notre recherche.
Par exemple si on compare le motif TTCACCGA et qu'on trouve une lettre T dans la chaîne mais pas à la bonne position, alors pour aligner la chaîne avec la lettre T, on devra effectuer un décalage de six lettres minimum.
Ce sera plus clair en utilisant d'abord un exemple plus simple que l'exemple précédent.
Supposons que l'on cherche le motif TOOTH dans le texte suivant : TRUSTHARDTOOTHBRUSHES
On commence par construire une table qui va nous donner les décalages à effectuer pour chaque lettre.
La lettre H est au début du mot est n'est pas présent ailleur dans le motif (il n'y a pas plusieurs fois cette lettre). Si ce caractère ne correspond pas, alors on va effectuer un décalage de 5 lettres où 5 correspond au nombre de lettres dans le motif.
Pour T, on commence à compter le nombre de décalage à partir de la dernière lettre. Dans ce cas on effectur un décalage d'une lettre , pour O un décalage de deux lettres. Dans ces deux cas on ne prend en compte que la première occurence de chaque lettre (on ne prend pas en compte les répétitions des lettres qui viennent ensuite).
Lorsqu'une lettre de la chaîne n'est pas présente dans le motif alors on effectue un décalage de 5 lettres.
On obtient alors la table de décalage suivante :
| Lettre | T | O | H | AUTRES LETTRES |
|---|---|---|---|---|
| Décalage | 1 | 2 | 5 | 5 |
TOOTH
TRUSTHARDTOOTHBRUSHES
On commence la comparaison entre le motif et la chaîne par la droite.
T ne correspond pas avec H, on effectue un décalage de une lettre (voir la table de décalage).
TOOTH
TRUSTHARDTOOTHBRUSHES
La lettre S n'est pas présente dans le motif, on effectue donc un décalage de 5 lettres.
TOOTH
TRUSTHARDTOOTHBRUSHES
La lettre O ne correspond pas, on effectue un décalage de deux lettres.
TOOTH
TRUSTHARDTOOTHBRUSHES
La lettre T ne correspond pas, on effectue un décalage de une lettre.
TOOTH
TRUSTHARDTOOTHBRUSHES
On compare chacune des lettres, elles correspondent toutes, on a alors trouvé le motif recherché dans la chaîne.
2.2) Exemple : ADN¶
Revenons maintenant à notre exemple précédent.
- On commence la comparaison entre le motif et la chaîne par la droite
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- La lettre A du motif correspond à la lettre A de la chaîne. La lettre G du motif ne correspond pas à la lettre A de la chaîne. On peut avancer de une lettre vers la droite.
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- Le A et le T ne correspondent pas. Pour faire correspondre le T avec la chaine nous allons décaler le motif de 6 lettres.
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- Le A et le T ne correspondent pas à nouveau, on décale alors à nouveau le motif de 6 lettres.
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- Cette fois-ci le G et le T ne correspondent pas, on décale à nouveau le motif de 6 lettres.
TTCACCGA
GTGCAGAATGAATTCTCGTAACTACATAGCACAAGTAGATGTAGTTAACTTTAATCTCACATAGCAATC
TTTAATC
AGTGTGTAACATTAGGGAGGACTTGAAAGAGCCACCACATTTTCACCGAGGCCACGCGGAGTAC
- Cette fois-ci le C ne correspond pas au G, on décale le motif de deux lettres pour aligner le C avec le texte.
On compare à nouveau chacune des lettres et on effectue les décalages qui conviennent jusqu'à ce qu'on ait trouvé le motif correspondant.
En TP vous implémenterez cet algorithme sur le génome d'un variant du SARS-CoV-2.